在合理区间内,Ct值小,感觉美滋滋,但Ct值大的时候,可能就难受了≧ ﹏ ≦
今天小翌就给大家分享一下如何应对Ct值偏大的问题~
Ct值,即循环阈值(Cycle Threshold),是指荧光定量PCR实验中荧光信号首次超过背景噪音水平时的循环次数,是最重要的关键参数,可用于分析基因表达差异、计算基因拷贝数或者实验质量控制等等。
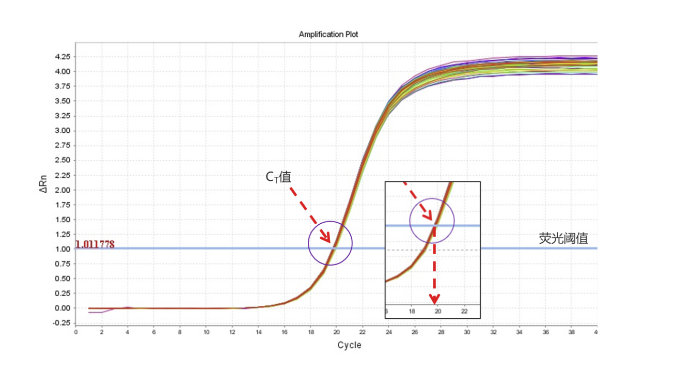
从图表上我们可以看到的是荧光阈值和扩增曲线交点的横坐标即为Ct值。其中出现的荧光阈值一般是基线的标准差的10倍,在实际操作中也可以手动调节。
而Ct值与模板量、产物量之间的关系为:
扩增产物量Xn=起始模板量X0×(1+扩增效率En)循环个数n
其中En表示的是扩增效率。当扩增产物量增长到一定量级后,所发出的荧光信号等于荧光阈值,此时的循环个数则为Ct值。由计算方式可以了解到,Ct值与起始模板的对数存在线性关系,当扩增产物量为定值时,起始模板量越大,Ct值越小;起始模板量越小,Ct值越大。
但是小伙伴们要注意,Ct值不是恒定不变的,不同的样本、不同的仪器会对该数据有一定的影响,有时即使是相同的样品在相同的仪器上重复2-3遍,Ct值也会存在差异。
头疼的问题 为什么会出现Ct值偏大的现象?
Ct值的范围通常在15-35之间。当Ct值小于15,则认为在基线期扩增范围内,未达到荧光阈值。当Ct值大于35时,不适宜用于定量,仅能做定性判断。但是有不少小伙伴表示,Ct值在30以上心里就很难受了,数据使起来不痛快,为什么会出现大的Ct值呢?
上文中有提到关系等式:
扩增产物量Xn=起始模板量X0×(1+扩增效率En)循环个数n
可以发现要达到一定的扩增产物量,Ct值同起始模板量和扩增效率En有一定的关系。
>起始模板量对于Ct值的影响
不同的小伙伴对于起始模板的投入量有不同的选择,例如选择将cDNA原液稀释10倍后作为反应模板上机检测;直接选择cDNA原液作为反应模板上机检测;又或者在进行反转录时选用100ng RNA或1000ng RNA进行反转录。

不同的反应模板量会对Ct值造成影响,模板量越多,Ct值越小,而模板量越少,Ct值就越大。10倍的模板量差异,对应的Ct值差异约在3.3个循环数(23.33=10)。
理论情况下,模板量投入越多,Ct值会变得越小,但实际操作时要注意!小伙伴们在阅读反转录试剂或者荧光定量试剂说明书时,会发现模板投入量都会存在上限。例如反转录时模板RNA投入量不建议超过2000ng,荧光定量PCR时投入的cDNA原液体积不能超过总反应体系的1/10。切记不要盲目为了减少Ct值无限制地增加模板投入量。模板本身除了包含核酸外,还有其他潜在的上游反应残留物或抑制物,量大时会对下游反应造成一定的抑制作用。

>扩增效率对于Ct值的影响
理论情况下,PCR扩增将1个DNA分子变成2个DNA分子,扩增效率为100%。但实际情况下,由于PCR反应中存在抑制因素,导致扩增效率低于100%,而一些污染、非特异扩增或者引物二聚体会导致扩增效率高于100%。
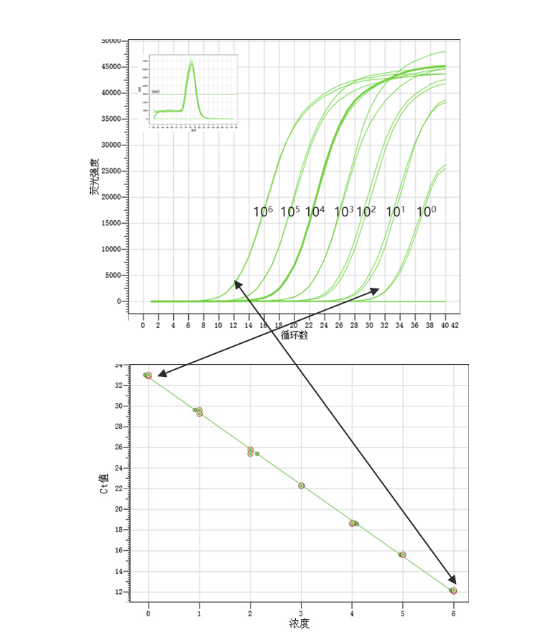
通常建议选用扩增效率在90%-110%之间的反应体系进行实验测试。看到这,小伙伴会问,我如何知道我的反应体系扩增效率在多少?
通常获取真实的扩增效率需要通过预实验制作标准曲线,通过制作标准曲线,获取曲线斜率,从而换算出扩增效率。
标准曲线制作案例:以染料法荧光定量检测A基因
01
尽量准备高浓度的待测cDNA或质粒DNA,于室温中瞬离混匀备用。根据个人需求按照一定梯度稀释核酸样本,一般建议6-8个梯度。
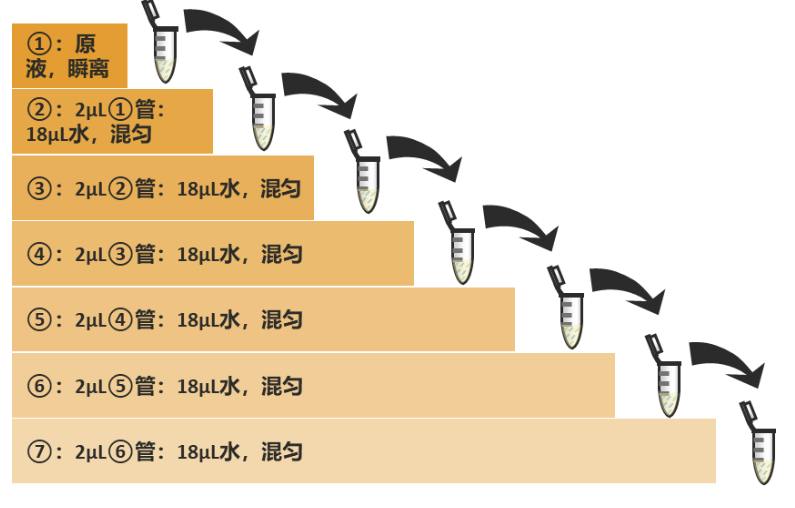
02
每个梯度做3个技术重复,配制大体系平均分配至小体系准确定会更高。期间也要注意混匀,枪头的替换和气泡排除等操作细节。
03
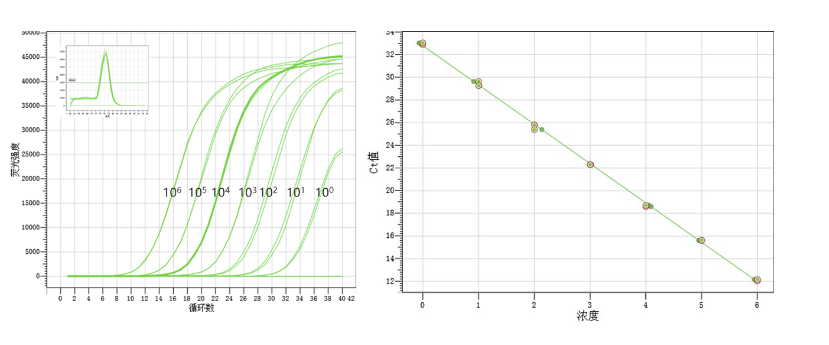
上机后,荧光定量PCR仪器会根据输入的信息进行标准曲线作图和换算。
04
观察标准曲线的斜率是否落在-3.58~-3.10之间以及相关系数R2是否≥0.98。(斜率折算成扩增效率的算法:扩增效率=10(-1/斜率)-1)
选用扩增效率在90%-110%之间的反应体系,当然,越接近100%越好。当扩增效率下降5%时,对应的Ct值大约会增加1。当扩增效率较低,在80%以下时,Ct值会明显偏大。
>出现Ct值大,该怎么办?
首先,小伙伴们,当Ct值偏大,尤其在30以上时,切记先不要慌,先保存好辛苦做出来的数据,冷静下来后细心分析下数据,看看数据本身是否合理且可使用!若复孔Ct值的STD<0.2且熔解曲线单峰,则表明该数据是可靠的,可用于数据分析。
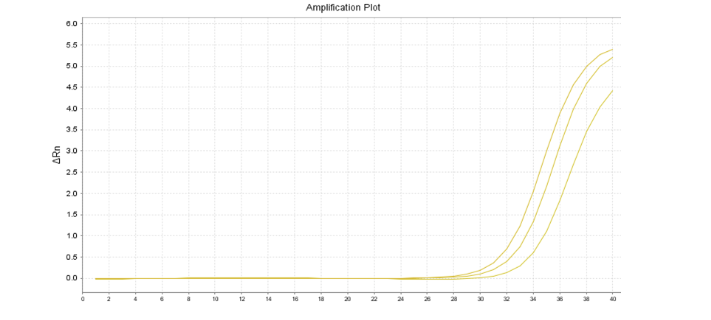
当Ct值大于30且复孔STD值>0.2时,若其本身为低表达基因且在其他研究或发表文献中都得到验证,可尝试将技术重复从3管提升至5-6管。再次上机后,剔除其中差异较大的技术重复,选用STD值最小的复孔数据。
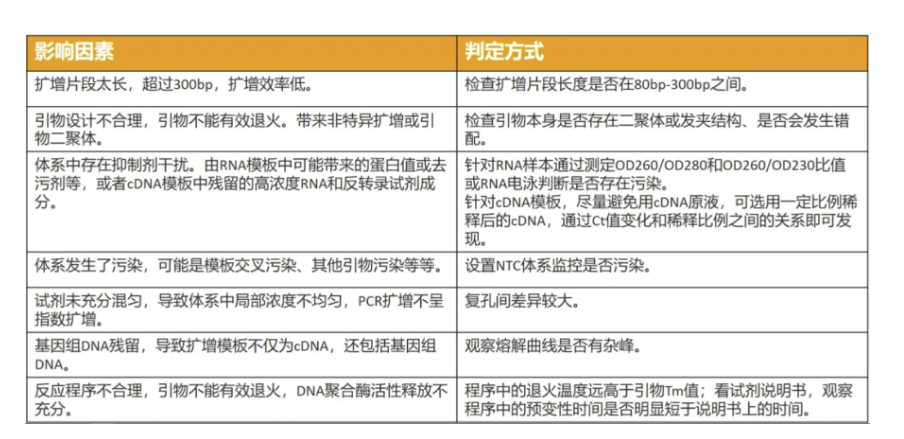
对症下药,减小Ct值 01
目标模板浓度偏低
a. 通过增加反转录时RNA的投入量从而提高模板浓度,或减少cDNA模板稀释度,理论上每稀释10倍,Ct值增大3.3。
b. 模板降解的风险,选用新鲜的RNA或分装保存好的未开封RNA进行反转录获取新的cDNA。选择高产高稳的RNA提取试剂及反转录试剂。
c. 若基因本身表达丰度就很低,尝试选用高灵敏的qPCR试剂来进行荧光定量PCR。
02
扩增片段过长
a.扩增产品片段不要过长,选择产物长度在80-300bp之间的。
b.可尝试标准的三步法扩增提高长片段的扩增效率。但是要注意片段长度很长的,该方法效果不大,建议选用方法a。
03
扩增效率异常,严重偏离100%±10%
a. 做好引物设计,避免二级结构、发夹结构和错配,减少非特异扩增。
b. 确保RNA纯度达标(OD260/280、OD260/230),减少抑制物对于反应的影响。
c. 若原采用cDNA原液作为模板,可尝试对cDNA原液进行一定比例稀释后进行分析。
种草推荐
目前翌圣qPCR mix系列的产品已经荣登Nature、 Cell等多个顶级期刊,获得科研大牛们认可!以下仅展示部分助力发表的高分文章:
[1] Li Y, Wang D, Ping X, et al. Local hyperthermia therapy induces browning of white fat and treats obesity. Cell. 2022;185(6):949-966.e19.doi:10.1016/j.cell.2022.02.004.(IF=66.850)
[2] Seki T, Yang Y, Sun X, et al. Brown-fat-mediated tumour suppression by cold-altered global metabolism. Nature. 2022;608(7922):421-428. doi:10.1038/s41586-022-05030-3. (IF=69.504)
[3] Chen P, Wang W, Liu R, et al. Olfactory sensory experience regulates gliomagenesis via neuronal IGF1. Nature. 2022;606(7914):550-556. doi:10.1038/s41586-022-04719-9.(IF=69.504)
[4] Dong W, Zhu Y, Chang H, et al. An SHR-SCR module specifies legume cortical cell fate to enable nodulation. Nature. 2021;589(7843):586-590. doi:10.1038/s41586-020-3016-z.(IF=69.504)
[5] Lu XY, Shi XJ, Hu A, et al. Feeding induces cholesterol biosynthesis via the mTORC1-USP20-HMGCR axis. Nature. 2020;588(7838):479-484. doi:10.1038/s41586-020-2928-y.(IF=69.504)
[6] Bi X, Wang K, Yang L, et al. Tracing the genetic footprints of vertebrate landing in non-teleost ray-finned fishes. Cell. 2021;184(5):1377-1391.e14. doi:10.1016/j.cell.2021.01.046.(IF=66.850)
[7] Liu S, Hua Y, Wang J, et al. RNA polymerase III is required for the repair of DNA double-strand breaks by homologous recombination. Cell. 2021;184(5):1314-1329.e10. doi:10.1016/j.cell.2021.01.048.(IF=66.850)
[8] Liu CX, Li X, Nan F, et al. Structure and Degradation of Circular RNAs Regulate PKR Activation in Innate Immunity. Cell. 2019;177(4):865-880.e21. doi:10.1016/j.cell.2019.03.046.(IF=66.850)
[9] Han X, Wang R, Zhou Y, et al. Mapping the Mouse Cell Atlas by Microwell-Seq. Cell. 2018;172(5):1091-1107.e17. doi:10.1016/j.cell.2018.02.001.(IF=66.850)
[10] Chai Q, Yu S, Zhong Y, et al. A bacterial phospholipid phosphatase inhibits host pyroptosis by hijacking ubiquitin. Science. 2022;378(6616):eabq0132. doi:10.1126/science.abq0132.(IF=63.714)
[11] Yu Q, Liu S, Yu L, et al. RNA demethylation increases the yield and biomass of rice and potato plants in field trials. Nat Biotechnol. 2021;39(12):1581-1588. doi:10.1038/s41587-021-00982-9.(IF=68.164)
[12] Han F, Liu X, Chen C, et al. Hypercholesterolemia risk-associated GPR146 is an orphan G-protein coupled receptor that regulates blood cholesterol levels in humans and mice. Cell Res. 2020;30(4):363-365. doi:10.1038/s41422-020-0303-z.(IF=46.297)
[13] Wang Z, Lu Z, Lin S, et al. Leucine-tRNA-synthase-2-expressing B cells contribute to colorectal cancer immunoevasion. Immunity. 2022;55(6):1067-1081.e8. doi:10.1016/j.immuni.2022.04.017.(IF=43.474)
[14] Bi Q, Wang C, Cheng G, et al. Microglia-derived PDGFB promotes neuronal potassium currents to suppress basal sympathetic tonicity and limit hypertension. Immunity. 2022;55(8):1466-1482.e9. doi:10.1016/j.immuni.2022.06.018.(IF=43.474)
[15] Wang X, Ni L, Wan S, et al. Febrile Temperature Critically Controls the Differentiation and Pathogenicity of T Helper 17 Cells. Immunity. 2020;52(2):328-341.e5. doi:10.1016/j.immuni.2020.01.006.(IF=43.474)
[16] Xiao J, Li W, Zheng X, et al. Targeting 7-Dehydrocholesterol Reductase Integrates Cholesterol Metabolism and IRF3 Activation to Eliminate Infection. Immunity. 2020;52(1):109-122.e6. doi:10.1016/j.immuni.2019.11.015.(IF=43.474)
[17] Zhang X, Zhang C, Qiao M, et al. Depletion of BATF in CAR-T cells enhances antitumor activity by inducing resistance against exhaustion and formation of central memory cells. Cancer Cell. 2022;40(11):1407-1422.e7. doi:10.1016/j.ccell.2022.09.013.(IF=38.585)
[18] Wang XY, Wei Y, Hu B, et al. c-Myc-driven glycolysis polarizes functional regulatory B cells that trigger pathogenic inflammatory responses. Signal Transduct Target Ther. 2022;7(1):105. Published 2022 Apr 18. doi:10.1038/s41392-022-00948-6.(IF=38.104)
[19] Fan H, Hong B, Luo Y, et al. The effect of whey protein on viral infection and replication of SARS-CoV-2 and pangolin coronavirus in vitro. Signal Transduct Target Ther. 2020;5(1):275. Published 2020 Nov 24. doi:10.1038/s41392-020-00408-z.(IF=38.104)
[20] Ren Y, Wang A, Wu D, et al. Dual inhibition of innate immunity and apoptosis by human cytomegalovirus protein UL37x1 enables efficient virus replication. Nat Microbiol. 2022;7(7):1041-1053. doi:10.1038/s41564-022-01136-6.(IF=30.964)
<上下滑动查看更多>

产品特别推荐
|
方法 |
分类 |
产品名称 |
货号 |
|
RNA提取 |
同Trizol提取 |
TRIeasy™ Total RNA Extraction Reagent |
|
|
免氯仿升级版 |
TRIeasy™ Total RNA Extraction Reagent(Tcm Free) |
||
|
动物组织/细胞总RNA提取,避开有毒试剂,最快15 min完成 |
MolPure® Cell/Tissue Total RNA Kit细胞/组织总RNA提取试剂盒 |
||
|
反转录试剂 |
5 min一步gDNA去除&反转录预混液(下游应用qPCR) |
Hifair® AdvanceFast One-step RT-gDNA Digestion SuperMix for qPCR |
|
|
5 min快速反转,最长可满足14 kb cDNA合成,含gDNA去除(下游应用PCR/qPCR) |
Hifair® AdvanceFast 1st Strand cDNA Synthesis Kit |
11149/11150ES |
|
|
高质量第一链cDNA合成预混液,含gDNA去除(下游应用qPCR) |
Hifair® III 1st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus) |
||
|
qPCR染料法 |
高特异高荧光值定量预混液(染料法) |
Hieff UNICON® Advanced qPCR SYBR Master Mix |
|
|
高灵敏通用型定量预混液(染料法) |
Hieff UNICON® Universal Blue qPCR SYBR Master Mix |
||
|
超高性价比定量预混液 (染料法),已发文章累计IF达到5000+ |
Hieff® qPCR SYBR Green Master Mix (No Rox) |
||
|
Hieff® qPCR SYBR Green Master Mix (Low Rox) |
|||
|
Hieff® qPCR SYBR Green Master Mix (High Rox) |
